Ficheros EDF de señales fisiológicas
Cuándo pretendemos aprender a trabajar con series temporales, resulta muy útil contar con buenos conjuntos de datos, y si son datos reales, mucho mejor. Resulta complicado conseguir series largas o que presenten patrones interesantes y bien localizados e identificados con las que podamos realizar prácticas. Una fuente excelente de series temporales complejas es nuestro propio organismo, y todo lo que podemos aprender trabajando con ellas es extrapolable a cualquier otro contexto.
Las señales obtenidas por electrocardiografía (ECG) o electroencefalografía (EEG) son quizás los ejemplos más conocidos, pero existen muchas otras posibilidades, como las series RR de intervalos entre latidos en un electrocardiograma, series con los niveles en sangre de glucosa, o cualquier otro marcador metabólico. Existen registros de señales que contienen horas de grabación, con miles de muestras, y colecciones de señales pertenecientes a distintos pacientes que presentan determinadas patologías.
Una excelente fuente de este tipo de datos es PhysioNet, que además es de acceso libre. Las señales contenidas en esta base de datos se pueden descargar como ficheros en diferentes formatos. En este artículo me voy a centrar en el formato EDF, que permite incluir toda una colección de señales para un mismo individuo. Este formato de archivo puede ser leído utilizando el entorno estadístico R, que es gratuito y se puede descargar e instalar fácilmente desde el sitio del Proyecto R. Este entorno proporciona un lenguaje de programación similar al C, y existen innumerables librerías para todo tipo de procedimientos estadísticos. También permite exportar datos en un gran número de formatos diferentes, por lo que resulta una excelente herramienta, ya sea para trabajar directamente con ella o bien para hacer de puente con cualquier otro sistema con el que trabajemos habitualmente.
No todos los archivos de la base de datos PhysioNet están disponibles en formato EDF, pero en el propio sitio existe una aplicación online para exportar cualquiera de los conjuntos de datos en este formato, se trata de PhysioBank ATM, y en este enlace tenéis una explicación exhaustiva sobre cómo utilizarla.
En cualquier caso, una vez que disponemos de un archivo en formato EDF, para cargarlo en el entorno R es necesario cargar la librería edfReader. Esta librería está disponible para su descarga utilizando la opción Instalar paquete(s) del menú Paquetes. Una vez instalada, podemos cargarla con el siguiente comando:
library(edfReader)
Vamos a descargar un fichero cualquiera que ya tenga este formato. Esta, por ejemplo, es una base de datos con señales procedentes de pacientes con apnea del sueño. Los archivos con extensión .rec y .edf tienen el formato que nos interesa. En la propia página tenéis una explicación sobre las señales contenidas en estos ficheros. Yo usaré ucddb003_lifecard.edf, que contiene tres de las señales que componen un electrocardiograma de larga duración (Holter).
Primero es necesario leer los datos de cabecera del fichero:
hdr<-readEdfHeader("ucddb003_lifecard.edf")
Y, a continuación, con esta cabecera, leer los datos:
sdata<-readEdfSignals(hdr)
De esta manera podemos ver cuáles son las señales contenidas en el archivo y algunos datos sobre ellas:
summary(sdata)
Start time : 2006-01-01 09:03:58
Ordinary signals:
Continuous recording : TRUE
Recorded period : 29100 sec = 08:05:00 h:m:s
Period read : whole recording
Signals
R/EDF name/label transducer sampleRate preFilter samples
signal /sec
1 chan 1 Ag-AgCl 128 … 3724800
2 chan 2 Ag-AgCl 128 … 3724800
3 chan 3 Ag-AgCl 128 … 3724800
Existen tres señales diferentes: chan 1, chan 2 y chan 3, correspondientes a tres de los canales del ECG. Se puede comprobar que la grabación ha durado 8 horas y 5 minutos y que cada señal contiene 3.724.800 muestras. Si observamos las primeras muestras de cualquiera de las señales, veremos simplemente una onda cuadrada que se utiliza para sincronizar el aparato de grabación con el centro de recogida de datos, pues se suele hacer a través de la línea telefónica:
plot(sdata$"chan 2"$signal[1:1000],type="l")
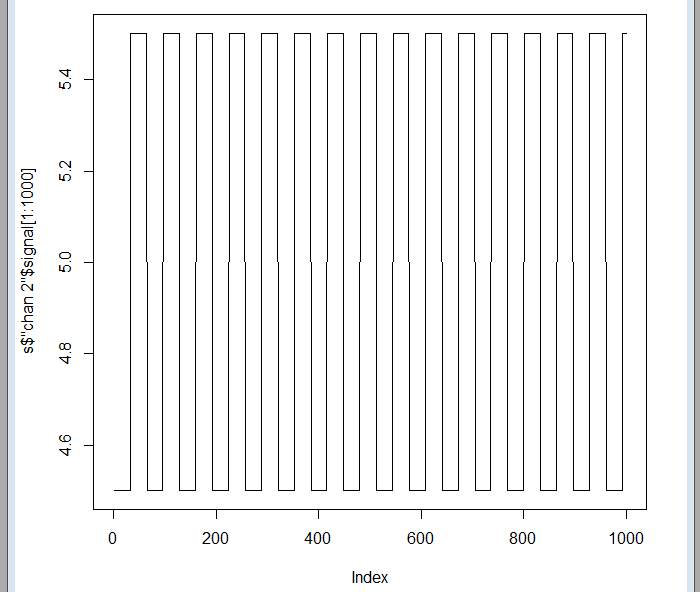
Pero algo más adelante ya se encuentran los datos:
plot(sdata$"chan 2"$signal[20000:21000],type="l")
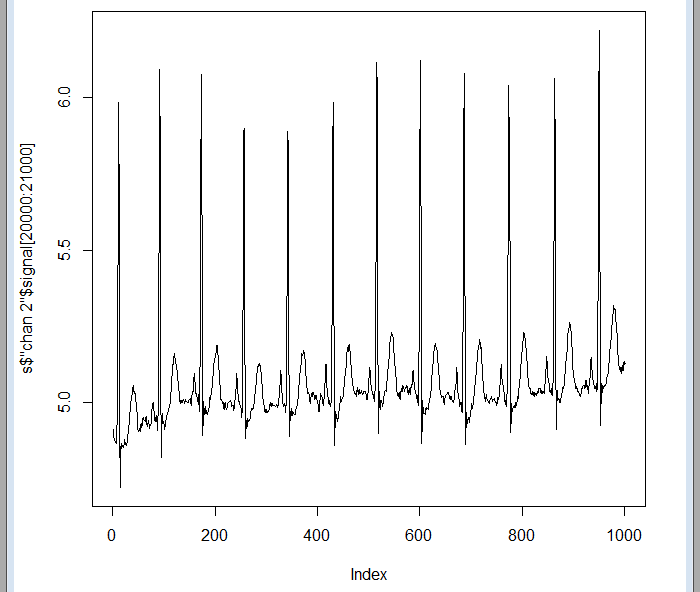
Como pasaría con cualquier otra fuente de datos que queramos analizar, es evidente que será necesario aprender algo sobre este tipo de señales para saber qué es lo que podemos encontrar en ellas y como son las diferentes irregularidades que podemos detectar o predecir, lo que para el caso de las señales fisiológicas se llamaría patologías. Esto ya corre por cuenta de cada uno, yo, por mi parte, iré escribiendo artículos de vez en cuando sobre formas de trabajar con este tipo de señales.









