Series temporales, RQA y redes neuronales II
En este segundo artículo de la serie sobre la utilización combinada del análisis de cuantificación de recurrencia (RQA) y las redes neuronales para trabajar con series temporales complejas, vamos a examinar algunas ideas sobre posibles tratamientos que se les pueden aplicar a los datos y a la selección de parámetros. Trabajaremos usando señales electrocardiográficas, según lo comentado en el anterior artículo.
En este enlace puedes acceder al primer artículo de esta serie, donde realizo una introducción general sobre el tema. Los ejemplos de datos y códigos que utilizaré los puedes descargar en este enlace.
Los electrocardiogramas que utilizaremos proceden de la base de datos PhysioNet de señales fisiológicas. Se trata de varios pares de electrocardiogramas procedentes de varios sujetos. Para cada uno de los sujetos, existe un electrocardiograma que corresponde a un estado normal sin actividad y otro tomado durante la realización de diferentes tareas de cálculo aritmético. Todos son sujetos sanos, y los datos de que disponemos sobre los individuos se encuentran recogidos en el archivo subject-info.xlsx.
El nombre de los archivos de datos de los sujetos se compone de la palabra Subject seguida de un número de dos cifras. Para diferenciar el electrocardiograma normal del correspondiente a las tareas aritméticas se utilizan los números 1 y 2, añadidos al final del nombre del archivo que contiene las series de tiempo. También disponemos de la edad, del género y del número de cálculos realizados correctamente durante el tiempo que duró la prueba, lo que proporciona una medida de la dificultad que les supuso completarla. Todas las series están grabadas utilizando la misma frecuencia de muestreo, por lo que no es necesaria modificación alguna en este sentido.
Los datos están, originalmente, en formato EDF, pero yo he grabado los que voy a utilizar en formato csv para simplificar su manejo. He seleccionado solo mujeres y solo los electrocardiogramas que corresponden a tareas de cálculo. El objetivo es comparar un grupo que ha obtenido buenos resultados con otro que los ha obtenido malos, y ver si los podemos diferenciar en base a los electrocardiogramas. También trataremos de aplicar las redes entrenadas de esta manera para identificar electrocardiogramas completos que no se hayan utilizado para el proceso de aprendizaje.
Puesto que se trata de sujetos sanos, la única suposición razonable a la hora de diferenciar un grupo del otro parece la de que la frecuencia cardíaca pueda ser diferente. El análisis de recurrencia nos proporciona información sobre la dinámica de una serie, y la dinámica del corazón no cambia así como así por realizar unos simples cálculos. Lo que sería esperable de este análisis es que los resultados no arrojasen diferencias entre ambos grupos, pero la cosa no es tan sencilla. Las redes neuronales son muy eficaces a la hora de encontrar diferencias en un conjunto de datos que pueda separarse en clases, incluso arbitrariamente, especialmente si dichas clases son linealmente separables. Por otra parte, RQA es bastante sensible a pequeñas diferencias inevitables entre latidos, incluso simplemente al ruido, por lo que aparecen diferencias espurias con mucha facilidad que pueden complicarnos bastante las cosas.
Preparando los datos
Empecemos por cargar las librerías y el código necesario para los ejemplos:
library(crqa)
library(RSNNS)
source(“ecgtools.r”)
El último archivo lo podéis encontrar entre los ejemplos de código y tiene algunas funciones útiles para trabajar con las señales. Al transformar una serie temporal en un conjunto de medidas RQA, una de las cosas que hay que decidir es la ventana de datos a utilizar, la longitud y posición de inicio de la porción o porciones de la serie. Cuanto más largas sean estas porciones, más tiempo llevará realizar el análisis, y los resultados serán diferentes con diferentes porciones y diferentes longitudes. En un electrocardiograma, parece natural utilizar el latido como unidad para dividir la serie en porciones. Podemos usar uno o más latidos; yo he optado por utilizar segmentos con un solo latido para disponer de una mayor cantidad de muestras.
El punto más apropiado para detectar el latido es el complejo QRS, en concreto, el pico R, por ser muy agudo. Aunque existen detectores de QRS y numerosos algoritmos más o menos complicados que podemos implementar, yo he preferido utilizar un método un tanto rudimentario pero que cumple con su función sin necesidad de grandes complicaciones: Los picos R son máximos, lo que significa que su segunda derivada es negativa. Como son picos muy pronunciados, el valor de la segunda derivada es relativamente alto, y esto lo podemos amplificar elevando su valor a una potencia impar, para que conserve el signo. Yo he realizado los cálculos con un valor de 5.
Así pues, calculamos la segunda derivada, la elevamos a la quinta potencia, descartamos todos los valores positivos y todos los negativos mayores que un determinado umbral, y nos deberían quedar solo los puntos que corresponden a los puntos R de la señal. El caso es que funciona bastante bien, siempre que el ECG no presente mucho ruido. En este caso, podemos realizar un filtrado previo de los datos para suavizar la señal o simplemente descartarla.
El problema es que este umbral hay que calcularlo visualmente, ya que no es el mismo para todos los electrocardiogramas, así que hay que realizar varios pasos. Como resultado, obtenemos una lista de las posiciones de todos los puntos R de la gráfica. Cada dos puntos consecutivos delimitan un latido. Voy a explicar gráficamente el proceso, aunque los cálculos están ya realizados en los ejemplos y guardados en los ficheros con extensión .int para cada uno de los ECGs correspondientes, almacenados en los archivos con extensión .csv.
signal<-read.csv2("Subject01_1.csv")[,1]
d25<-deriv2(signal)^5
plot(d25,type="l")
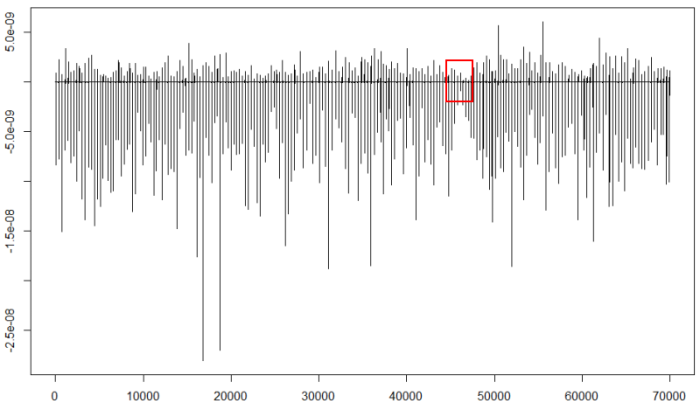
Nos fijamos en la parte que contenga los mínimos menos negativos (recuadro rojo) y la ampliamos para calcular el umbral:
plot(d25[46000:47000],type="l")
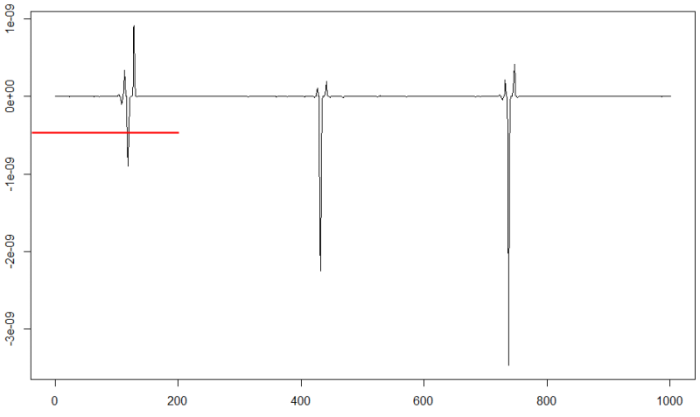
Probamos el valor umbral para comprobar si la señal se divide correctamente:
fil<-filterecg(d25,-0.5e-9)
plot(fil,type="l")
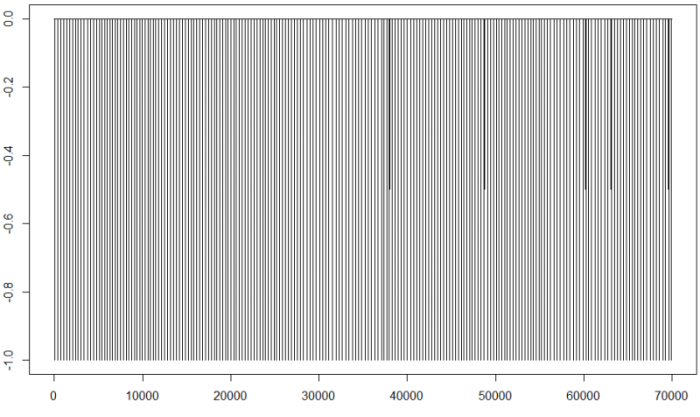
Donde solo existen valores a 0 o a -1 (para los puntos R). Si no observamos nada extraño, podemos pasar a calcular los intervalos para los latidos:
beats<-findr(signal,-0.5e-9)
plot(signal[beats[1,1]:beats[1,2]],type="l")
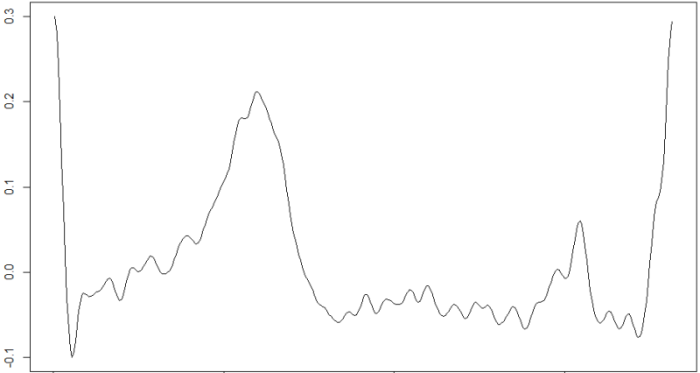
Donde beats es un array en el que cada fila es un latido, que empieza en la posición contenida en la primera columna y termina en la que contiene la segunda. Estos datos son los que se graban en los ficheros con extensión int asociados a cada csv con la señal del ECG.
Cálculo de los valores RQA
Una vez que podemos dividir la señal del electrocardiograma en latidos, tenemos una base para realizar las medidas RQA con una cierta homogeneidad. Yo voy a utilizar como unidad la señal entre dos puntos R consecutivos, pero esto es un criterio arbitrario, se podrían utilizar dos latidos o más como unidad, o seleccionar un segmento de tal manera que el punto R quedase en su interior. Esta es una de las cuestiones que uno debe decidir, porque los resultados obtenidos pueden variar bastante. En general, cuanta más longitud de señal procesemos, más tiempo tardarán los cálculos, y menos muestras tendremos a partir de una misma señal. También puede ser necesario escalar las señales para que sus rangos de valores no sean excesivamente diferentes. Yo he optado por escalar todos los ECG en un rango de valores entre -1.0 y 1.0.
La selección de parámetros incluye la dimensión embebida, el retardo y la distancia o radio, como expliqué en el primer artículo de esta serie. Para los dos primeros he elegido los valores 2 y 15. Estos valores provienen de algunos estudios que he consultado, en los que se aconseja extender el sistema a dos dimensiones y se utiliza un retardo de 0.03 segundos, que, con una frecuencia de muestreo de 500, corresponde a 15 puntos de la serie. La distancia o radio para considerar dos puntos como recurrentes la calculo en cada latido en función de uno de los valores RQA, la proporción de puntos recurrentes RR. El objetivo es obtener un valor similar para todas las muestras, de aproximadamente 1,25. La razón es que este parámetro se toma en ocasiones como referencia para calcular la distancia óptima, ya que los resultados varían mucho en función de dicha distancia. Se recomienda obtener valores no demasiado altos para el valor RR. Este es un mapa de recurrencia de un latido y los valores RQA usando una distancia de 0,099:
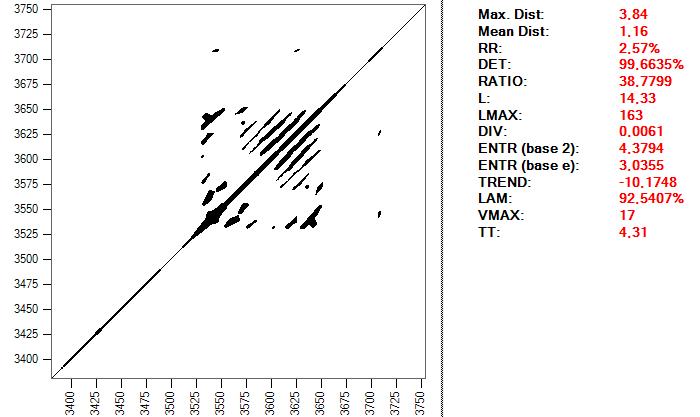
Y este es el mismo latido, pero usando una distancia de 0,2:
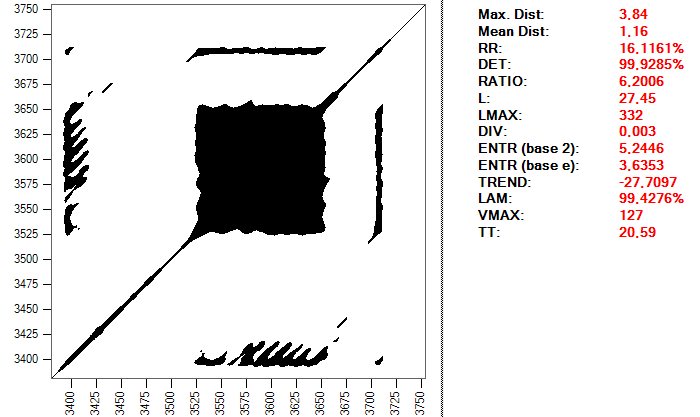
Lo importante es darse cuenta de la complejidad que puede encerrar simplemente realizar una buena elección de los parámetros que vamos a utilizar para realizar el estudio. Se trata de un proceso que puede llevar mucho tiempo y requerir muchas pruebas, además de un buen nivel de conocimiento sobre la materia y las herramientas utilizadas, no se trata precisamente de un procedimiento trivial.
El código que realiza los cálculos RQA para un segmento del ECG es la función mrqa, que a su vez utiliza la función rqa del paquete crqa:
mrqa<-function(data,clsval,edim=3,sdel=1,rad,rr=0) {
tmprad<- rad;
repeat {
rqa<-crqa(data,
data,
delay=sdel,
embed=edim,
radius=tmprad,
normalize=0,
rescale=0,
mindiagline=2,
minvertline=2,
side="lower");
if (rr == 0) {
break;
}
if (rqa$RR-rr < -0.05) {
tmprad<-tmprad+0.0001;
}
else if (rqa$RR-rr > 0.05) {
tmprad<-tmprad-0.0001;
}
else {
break;
}
}
vrqa<-c(rqa$RR,rqa$DET,rqa$NRLINE,rqa$maxL,
rqa$L,rqa$ENTR,rqa$rENTR,rqa$LAM,rqa$TT,clsval);
return(vrqa);
}
Para cada latido, se utiliza esta función para realizar los cálculos de análisis de recurrencia, y se genera un registro al que se le añade un identificador numérico de clase para entrenar posteriormente las redes neuronales. El parámetro data contiene el segmento de la señal; clsval es el valor numérico identificador de la clase asignada al ECG; edim es la dimensión embebida, sdel es el retardo a utilizar, rad es la distancia o radio, y rr es un parámetro opcional para ajustar la distancia de manera que los cálculos den como resultado un valor de la medida RR lo más aproximado posible a éste. Si se pasa el valor 0, no se realiza ningún ajuste. Hay que tener en cuenta que este ajuste puede hacer que los cálculos lleven mucho más tiempo.
Con la función rqadata se procesa un ECG completo:
rqadata<-function(vrqa,signalf,intf,clsval,edim=3,sdel=1,rad,
emin=-1,emax=1,rr=0) {
dint<-read.csv2(intf);
dsig<-read.csv2(signalf);
dnorm<-scaleecg(dsig[,1],emin,emax);
for(j in 1:length(dint[,1])) {
rqa<-mrqa(dnorm[dint[j,1]:dint[j,2]],clsval,edim,
sdel,rad,emin,emax,rr);
if (is.null(vrqa)) {
vrqa<-rqa;
}
else {
vrqa<-rbind(vrqa,rqa);
}
}
return(vrqa);
}
El parámetro vrqa contiene un dataframe con cálculos anteriores para añadirle nuevas filas, o bien NULL para comenzar un juego de datos nuevo; signalf contiene el nombre de un fichero csv con los datos de la señal del ECG; intf es el fichero con extensión int asociado al ECG y que contiene los índices de los latidos; emin y emax se utilizan para escalar los valores de la señal, y el resto de parámetros son los mismos utilizados en la función mrqa anterior.
En el próximo artículo, entrenaremos redes neuronales con los datos obtenidos mediante esta función.









